Datos de muestra
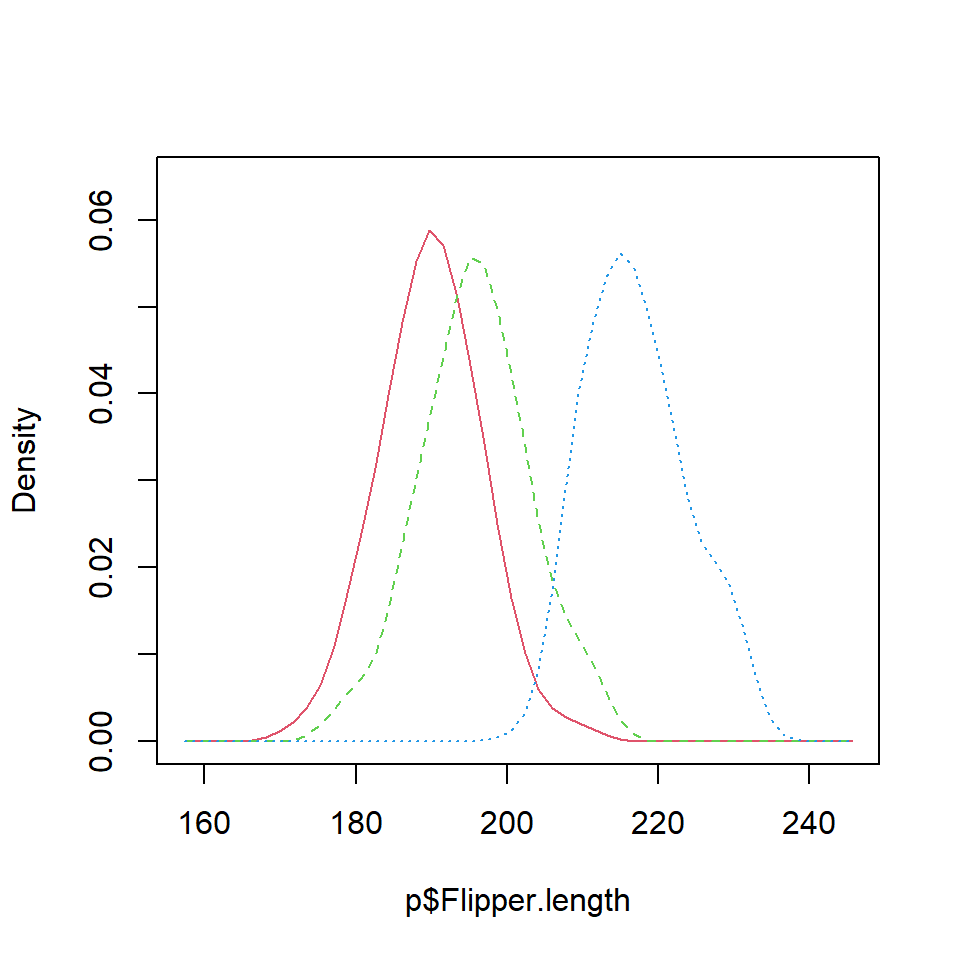
Considera los siguientes datos de la librería palmerpenguins, donde la variable continua es la longitud de las aletas de los pingüinos y la categórica la especie.
# install.packages("palmerpenguins")
library(palmerpenguins)
x <- penguins$flipper_length_mm
g <- as.factor(penguins$species)
p <- data.frame(Flipper.length = x, species = g)
p <- na.omit(p)
Comparación de densidades con lines
Es posible estimar las densidades tipo núcleo para cada especie con la función tapply. Luego, puedes añadir las tres densidades al mismo gráfico. Ten en cuenta que puedes que puedes seleccionar un kernel o una ventana distinta distintos a los valores por defecto.
# install.packages("car")
library(car)
# Densidades para cada grupo
dens <- tapply(p$Flipper.length, p$species, density)
# Gráfico
plot(dens$Adelie, xlim = c(min(p$Flipper.length) - 10,
max(p$Flipper.length) + 25),
main = "Comparación de densidades")
lines(dens$Chinstrap, col = 2)
lines(dens$Gentoo, col = 3)
# Leyenda
legend("topright", legend = levels(p$species),
lty = 1, col = 1:3)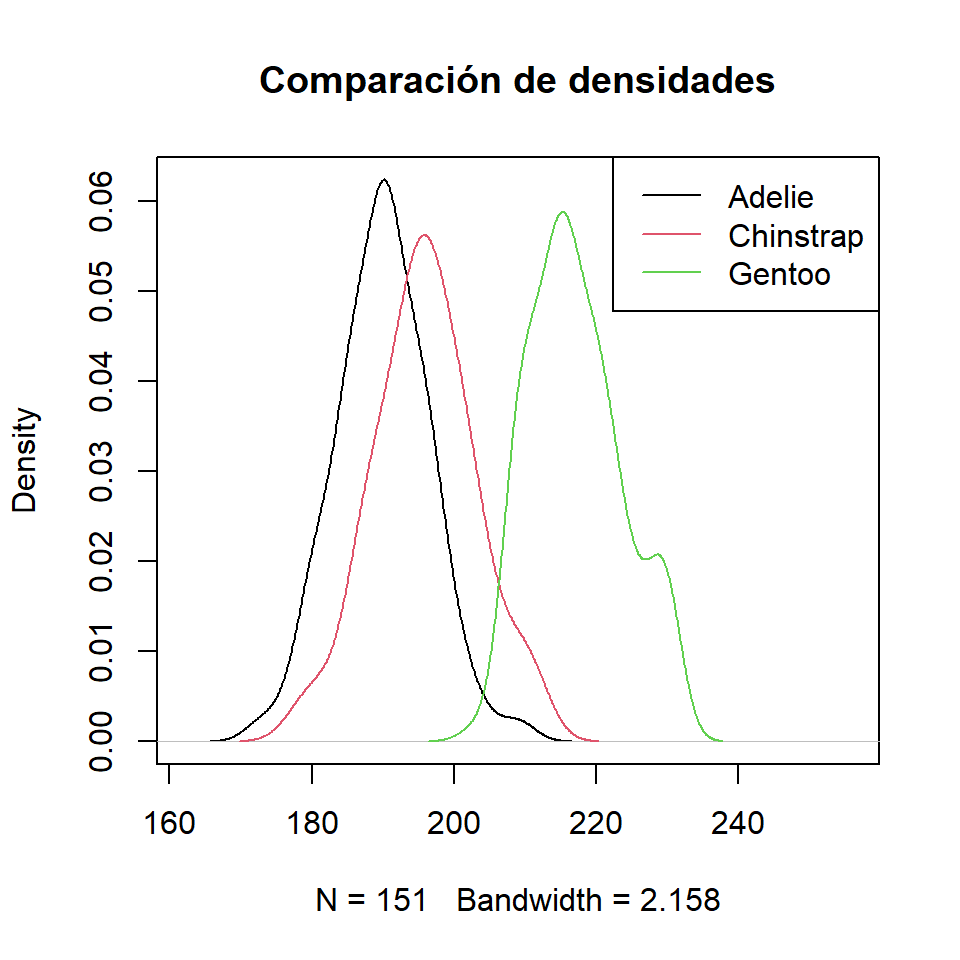
La función densityPlot
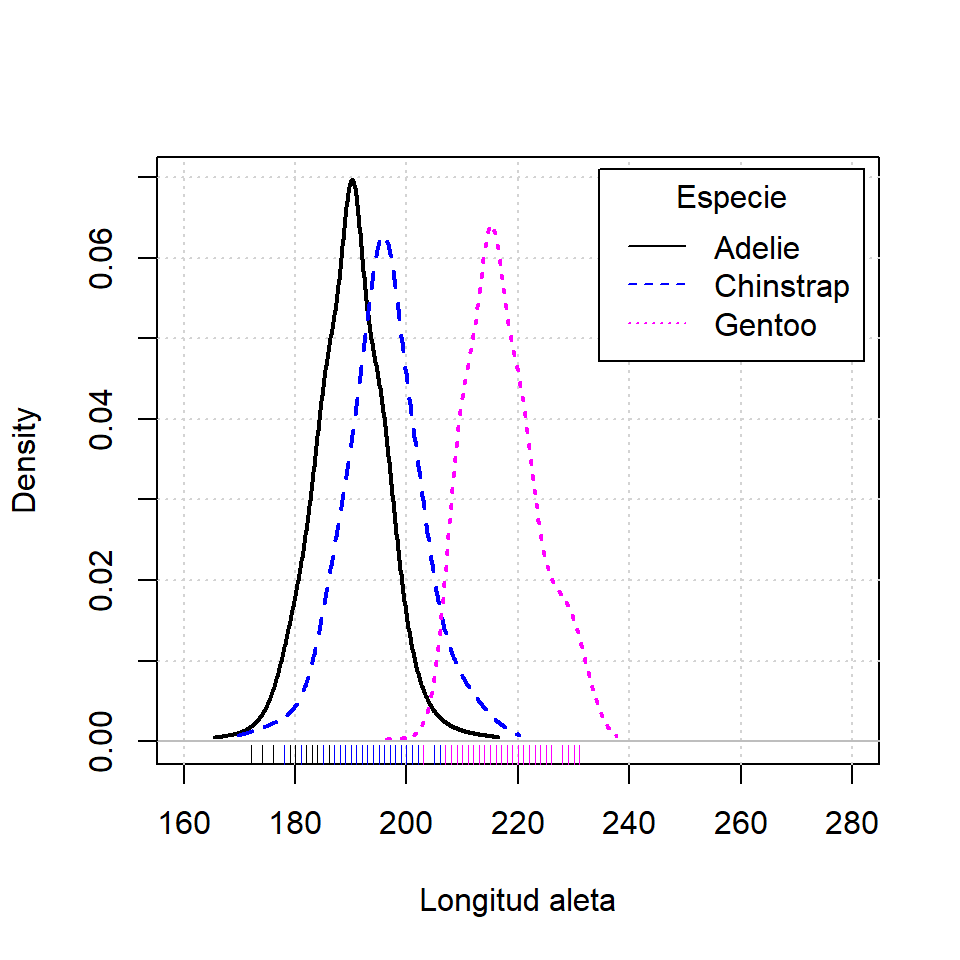
Una alternativa es usar la función densityPlot de la librería car. Ten en cuenta que esta función utiliza un kernel adaptativo por defecto en lugar de una estimación tipo núcleo con ventana fija. Si quieres obtener las mismas estimaciones que en la sección anterior pasa method = "kernel" como argumento.
# install.packages("car")
library(car)
densityPlot(p$Flipper.length ~ p$species,
legend = list(title = "Especie"),
xlab = "Longitud aleta",
xlim = c(160, 280))
La función sm.density.compare
Otra opción es utilizar la función sm.density.compare de la librería sm. Si estableces model = "equal" se ejecutará un contraste de permutaciones para la igualdad de densidades y se mostrarán intervalos de confianza bootstrap si el número de grupos es dos.
# install.packages("sm")
library(sm)
sm.density.compare(x = p$Flipper.length,
group = p$species,
model = "equal")