La función pheatmap
La función pheatmap es similar a la función heatmap de R base, pero proporciona más control sobre el gráfico resultante. Puedes pasar una matriz numérica que contenga los valores a ser representados.
# install.packages("pheatmap")
library(pheatmap)
# Datos
set.seed(8)
m <- matrix(rnorm(200), 10, 10)
colnames(m) <- paste("Col.", 1:10)
rownames(m) <- paste("Fila", 1:10)
# Mapa calor
pheatmap(m)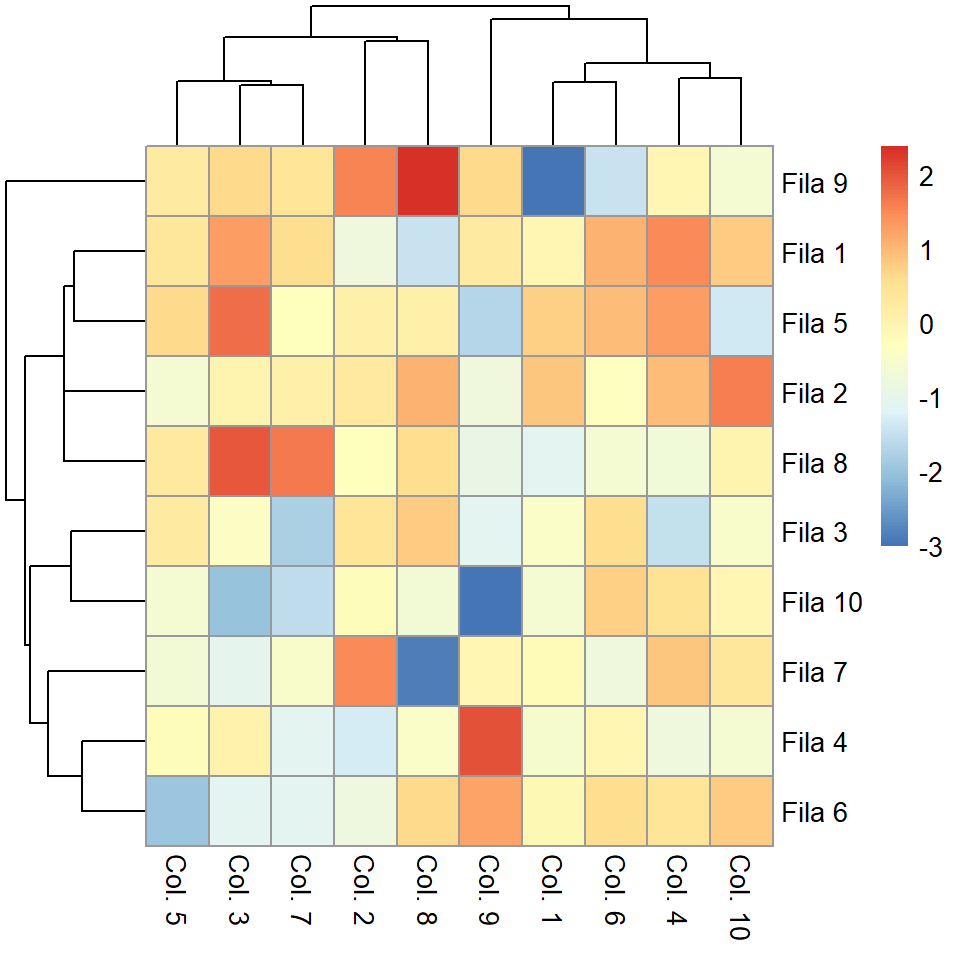
Normalización
Si los valores de la matriz no están normalizados puedes normalizar por filas ("row") o por columnas ("column") haciendo uso del argumento scale.
# install.packages("pheatmap")
library(pheatmap)
# Datos
set.seed(8)
m <- matrix(rnorm(200), 10, 10)
colnames(m) <- paste("Col.", 1:10)
rownames(m) <- paste("Fila", 1:10)
# Mapa calor
pheatmap(m, scale = "column")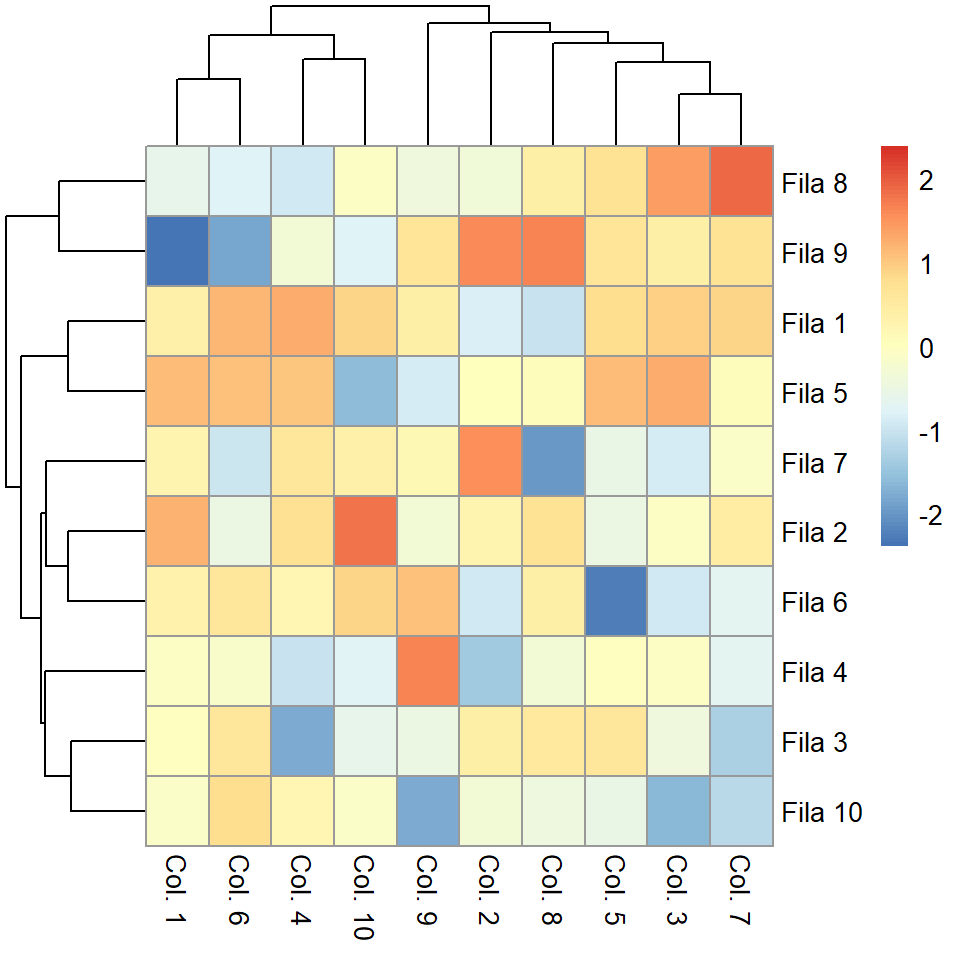
Valores
Si estableces display_numbers = TRUE se mostrarán los valores para cada celda. Tambén puedes especificar el color y el tamaño de los textos.
# install.packages("pheatmap")
library(pheatmap)
# Datos
set.seed(8)
m <- matrix(rnorm(200), 10, 10)
colnames(m) <- paste("Col.", 1:10)
rownames(m) <- paste("Fila", 1:10)
# Mapa calor
pheatmap(m,
display_numbers = TRUE,
number_color = "black",
fontsize_number = 8)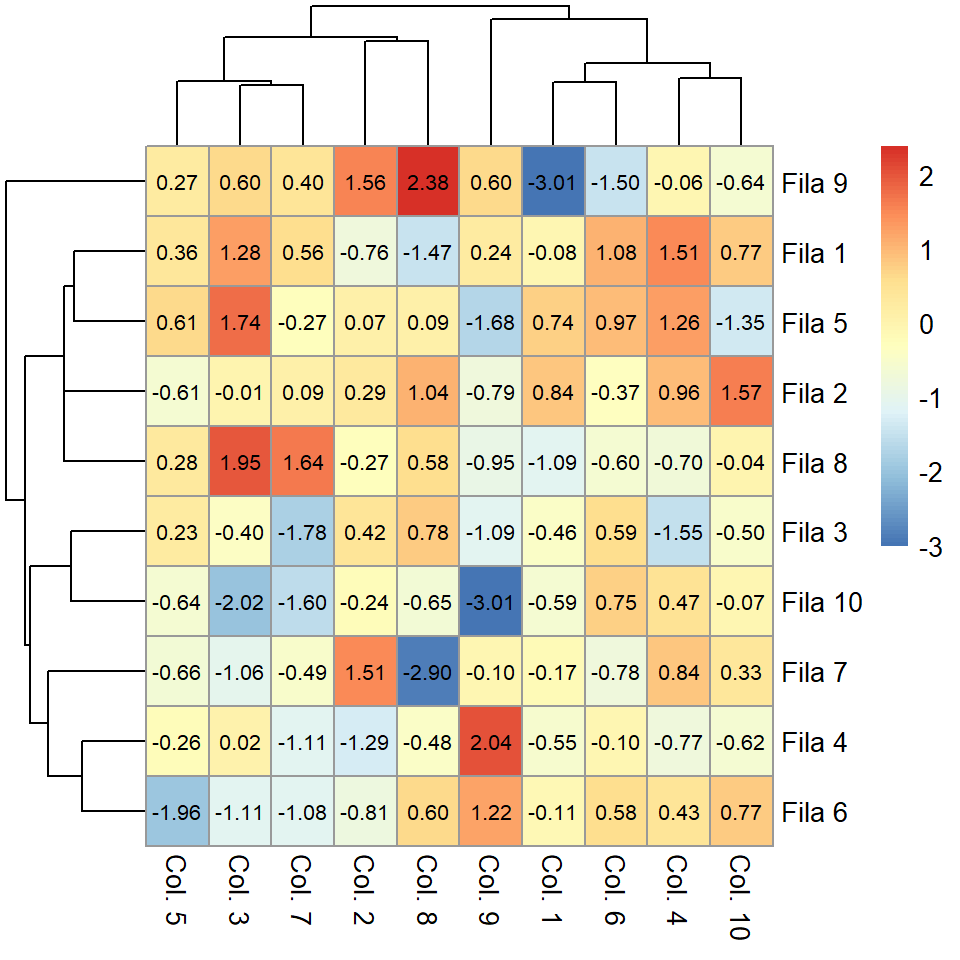
Número de clusters
El número de clusters (grupos) se puede cambiar con kmeans_k. Si el número de clusters es pequeño puedes incrementar el tamaño de las celdas con cellheight o cellwidth.
# install.packages("pheatmap")
library(pheatmap)
# Datos
set.seed(8)
m <- matrix(rnorm(200), 10, 10)
colnames(m) <- paste("Col.", 1:10)
rownames(m) <- paste("Fila", 1:10)
# Mapa calor
pheatmap(m, kmeans_k = 3, cellheight = 50)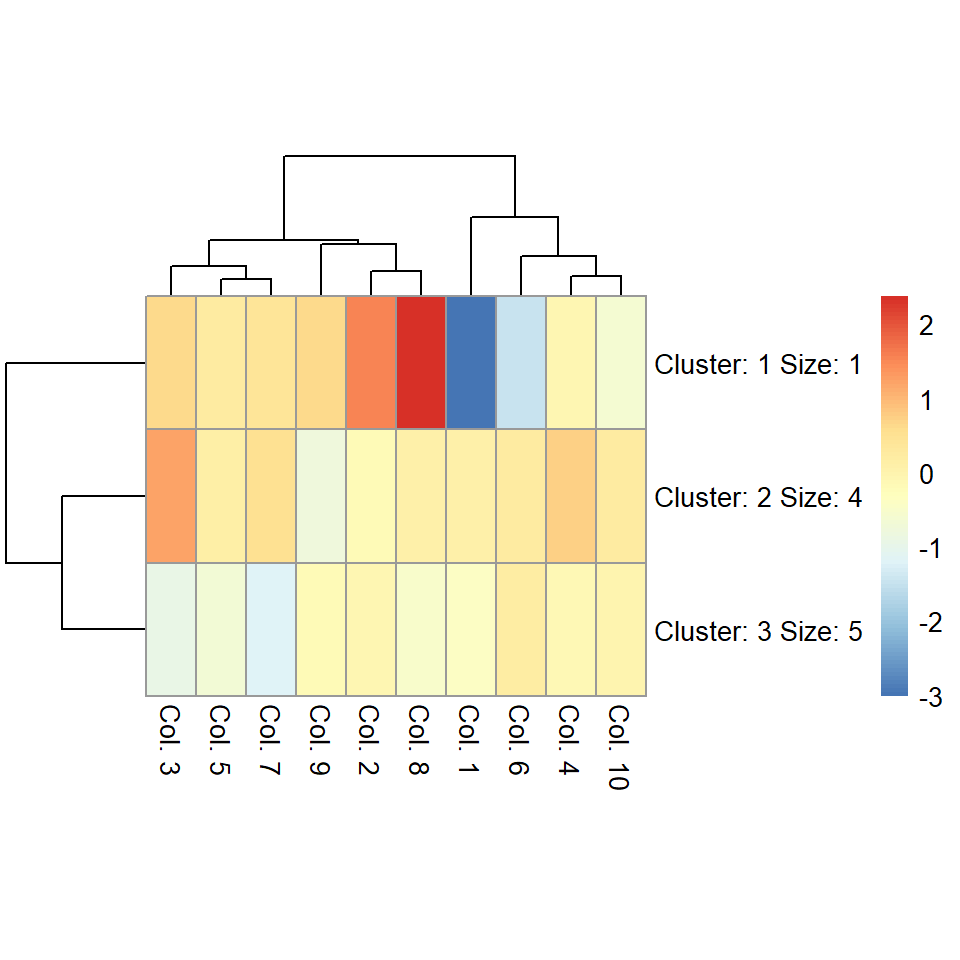
Dendrogramas
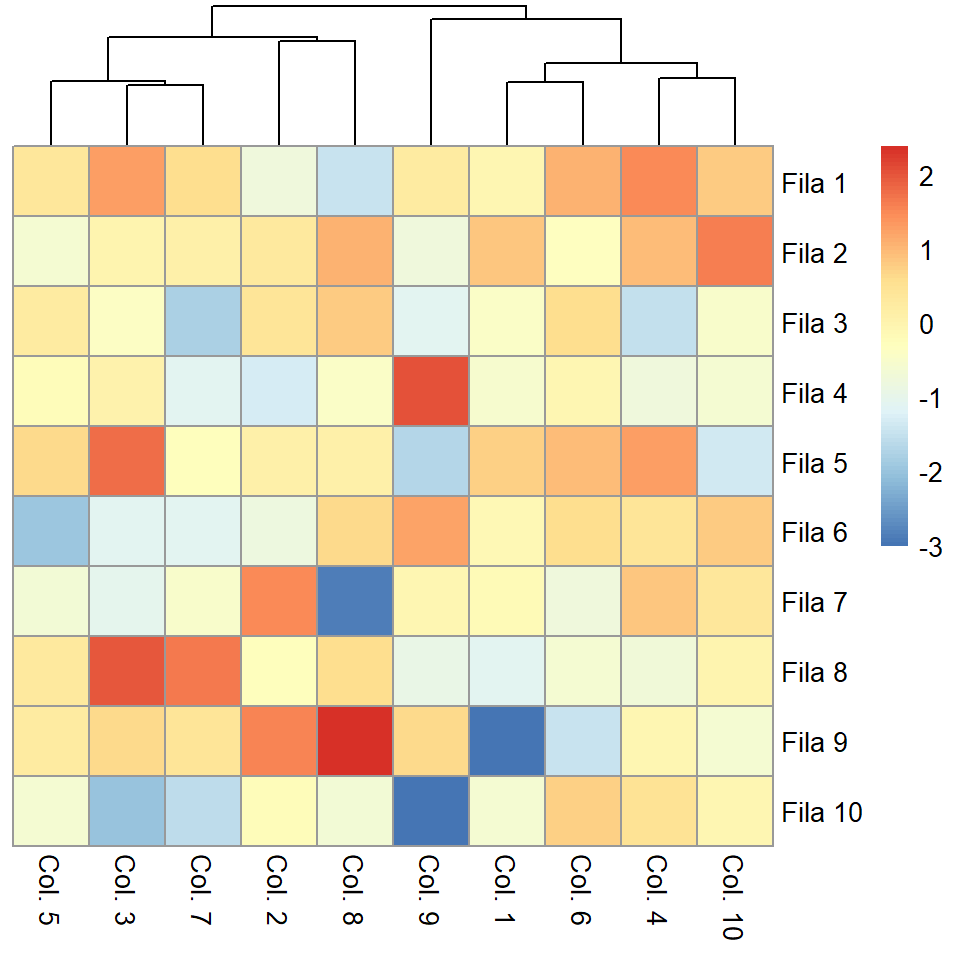
Eliminar el dendrograma de las filas
Puedes pasar un objeto hclust al argumento cluster_rows o establecerlo como FALSE para eliminar el dendrograma de las filas.
# install.packages("pheatmap")
library(pheatmap)
# Datos
set.seed(8)
m <- matrix(rnorm(200), 10, 10)
colnames(m) <- paste("Col.", 1:10)
rownames(m) <- paste("Fila", 1:10)
# Mapa calor
pheatmap(m, cluster_rows = FALSE)
Eliminar el dendrograma de las columnas
De manera equivalente al argumento anterior, cluster_cols controla cómo se debería crear el dendrograma de las columnas, en caso de crearse.
# install.packages("pheatmap")
library(pheatmap)
# Datos
set.seed(8)
m <- matrix(rnorm(200), 10, 10)
colnames(m) <- paste("Col.", 1:10)
rownames(m) <- paste("Fila", 1:10)
# Mapa calor
pheatmap(m, cluster_cols = FALSE)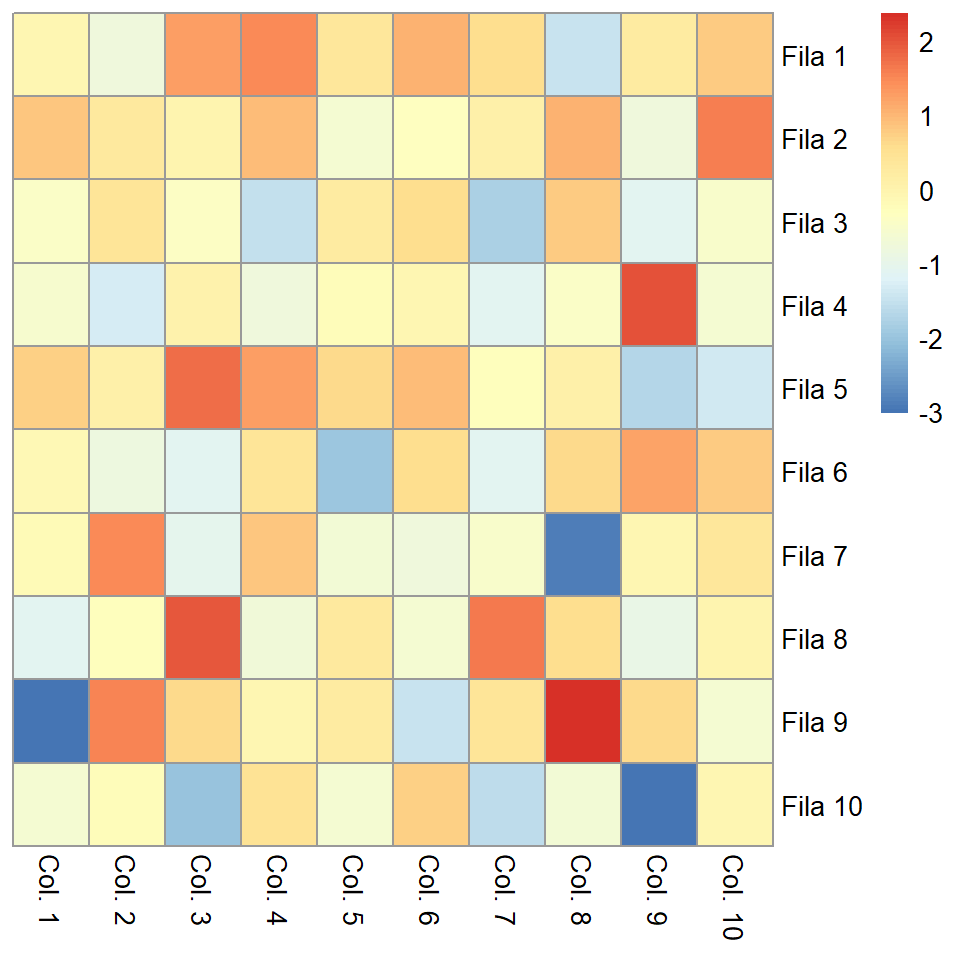
Eliminar los dendrogramas
Es posible eliminar ambos dendrogramas estableciendo cluster_cols y cluster_rows como FALSE.
# install.packages("pheatmap")
library(pheatmap)
# Datos
set.seed(8)
m <- matrix(rnorm(200), 10, 10)
colnames(m) <- paste("Col.", 1:10)
rownames(m) <- paste("Fila", 1:10)
# Mapa calor
pheatmap(m,
cluster_cols = FALSE,
cluster_rows = FALSE)Personalización del color
Color del borde
El argumento border_color controla el color del borde de las celdas. El valor por defecto es "grey60".
# install.packages("pheatmap")
library(pheatmap)
# Datos
set.seed(8)
m <- matrix(rnorm(200), 10, 10)
colnames(m) <- paste("Col.", 1:10)
rownames(m) <- paste("Fila", 1:10)
# Mapa calor
pheatmap(m, border_color = "black")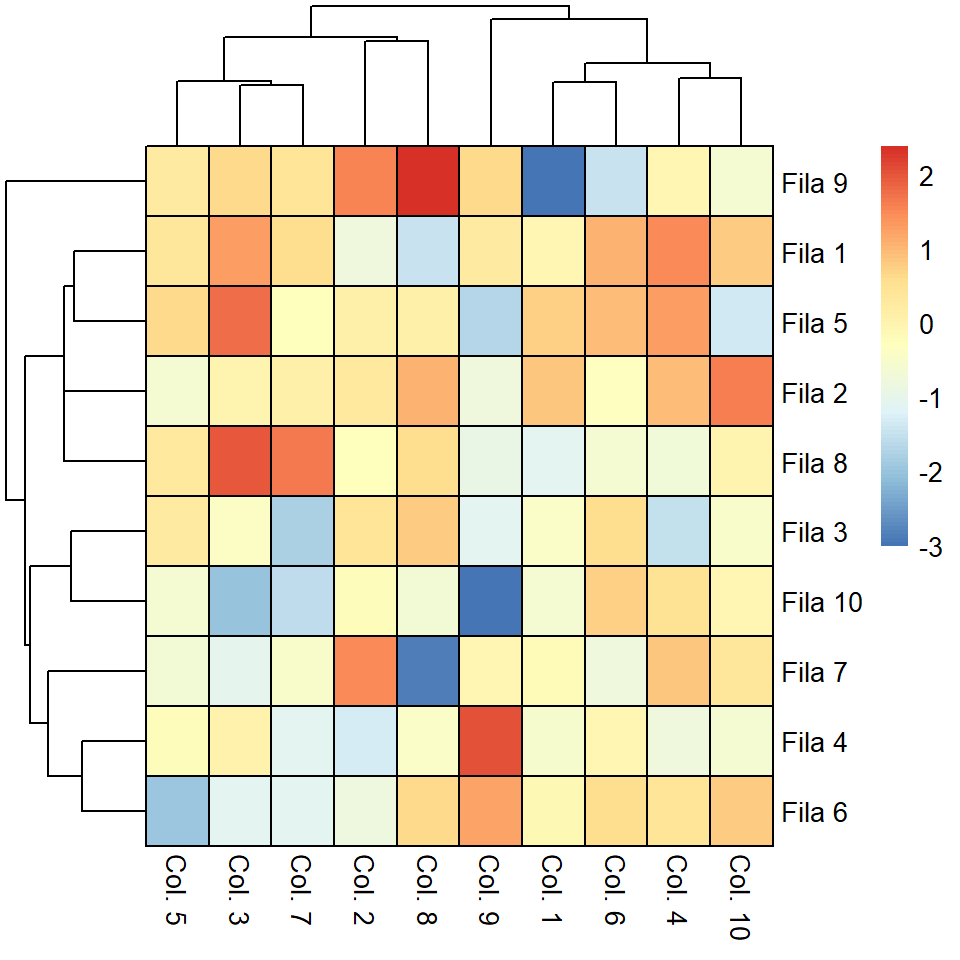
Paleta de colores
La paleta de colores por defecto se puede cambiar pasando un vector de colores al argumento color, tal y como se muestra en el siguiente ejemplo.
# install.packages("pheatmap")
library(pheatmap)
# Datos
set.seed(8)
m <- matrix(rnorm(200), 10, 10)
colnames(m) <- paste("Col.", 1:10)
rownames(m) <- paste("Fila", 1:10)
# Mapa calor
pheatmap(m, color = hcl.colors(50, "BluYl"))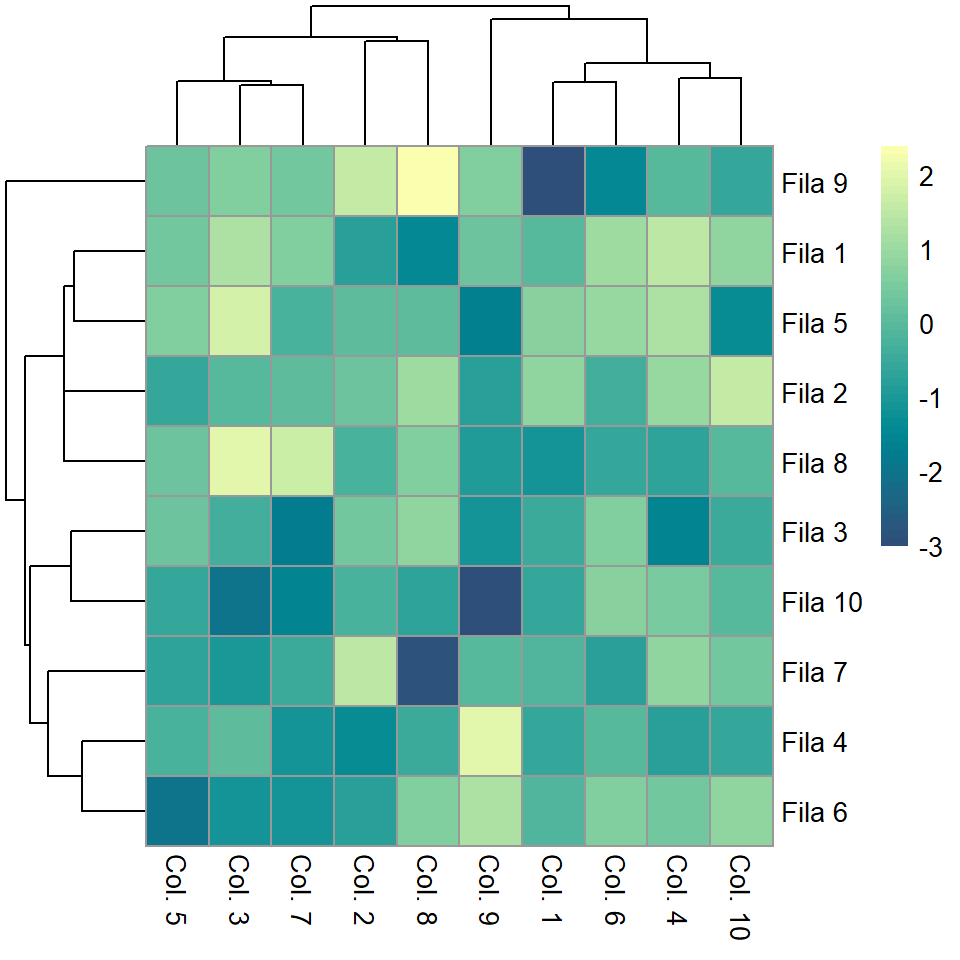
Personalización de la leyenda
Puntos de corte de la leyenda
El número de clases en los que se divide la leyenda se puede personalizar con legend_breaks, pasando los puntos de corte como vector.
# install.packages("pheatmap")
library(pheatmap)
# Datos
set.seed(8)
m <- matrix(rnorm(200), 10, 10)
colnames(m) <- paste("Col.", 1:10)
rownames(m) <- paste("Fila", 1:10)
# Mapa calor
pheatmap(m, legend_breaks = c(-2, 0, 2))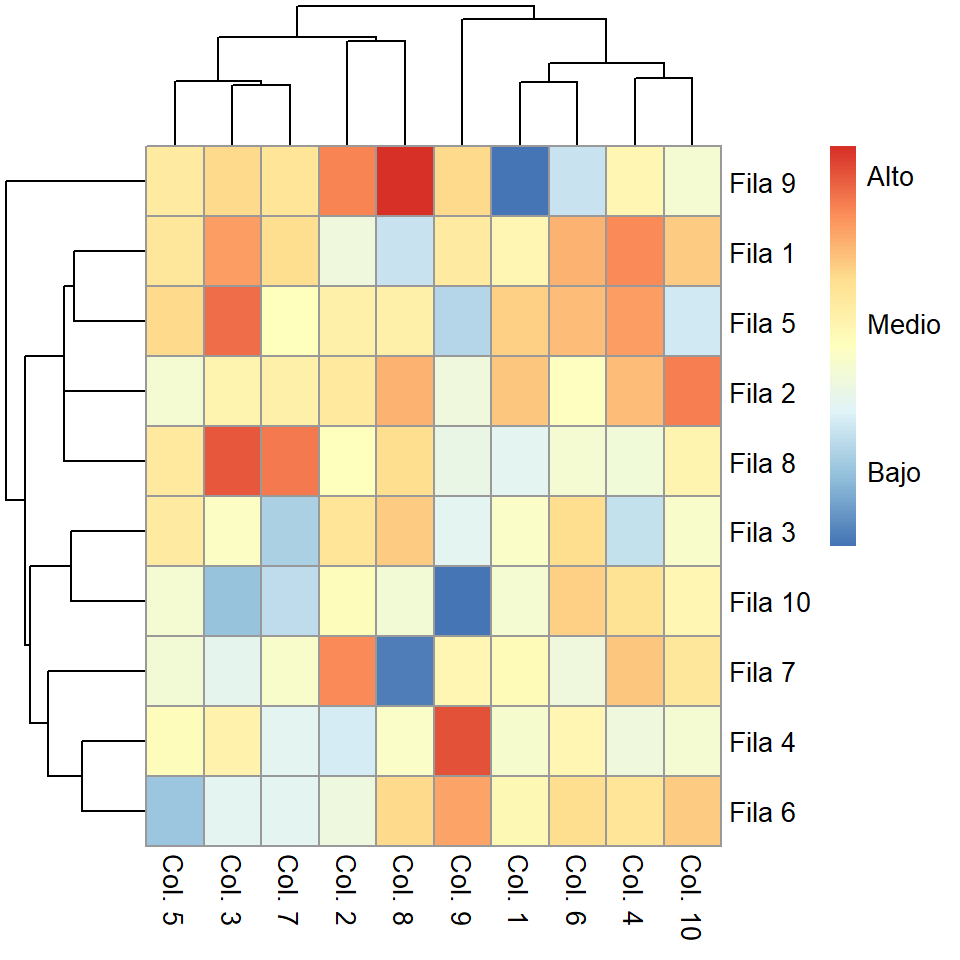
Etiquetas de la leyenda
Si estableces los puntos de corte también puedes pasar un vector de la misma longitud al argumento legend_labels para modificar las etiquetas.
# install.packages("pheatmap")
library(pheatmap)
# Datos
set.seed(8)
m <- matrix(rnorm(200), 10, 10)
colnames(m) <- paste("Col.", 1:10)
rownames(m) <- paste("Fila", 1:10)
# Mapa calor
pheatmap(m,
legend_breaks = c(-2, 0, 2),
legend_labels = c("Bajo", "Medio", "Alto"))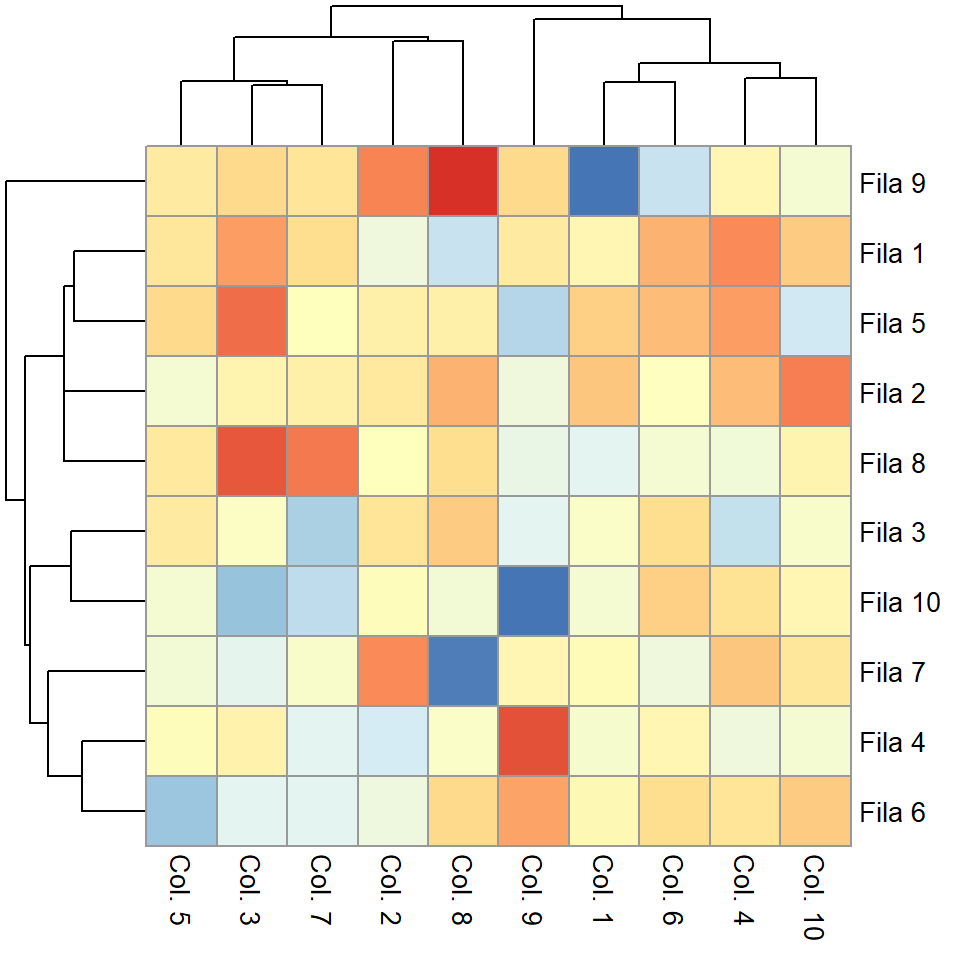
Eliminar la leyenda
Por último, si quieres deshacerte de la leyenda establece legend = FALSE.
# install.packages("pheatmap")
library(pheatmap)
# Datos
set.seed(8)
m <- matrix(rnorm(200), 10, 10)
colnames(m) <- paste("Col.", 1:10)
rownames(m) <- paste("Fila", 1:10)
# Mapa calor
pheatmap(m, legend = FALSE)